6.3 Mezclas gaussianas
Un caso más general es suponer que hay posibles clases y las distribuciones de la mezcla son normal multivariadas. De tal manera que
Entonces, la distribución de los datos observados es la mezcla Gaussiana:
donde es normal multivariada con parámetros ,
La estimación en el caso multivariado con clases se realiza con el algoritmo EM de manera análoga al caso de dos clases y normal univariada; sin embargo, es posible restringir el tipo de mezcla a clases de distribuciones Gaussianas determinadas por la matriz de covarianzas.
Consideremos la descomposición de espectral de la -ésima matriz de covarianzas:
es una matriz diagonal con y entradas diagonales proporcionales a los eigenvalores de . controla la forma (shape) del -ésimo cluster.
es una constante igual al que controla el volumen del -ésimo cluster.
es la matriz ortonormal de eigenvectores y controla la orientación del -ésimo cluster.
Utilizando la descomposición de arriba hay modelos de mezclas Gaussianas que se restringen a cluster con propiedades particulares, por ejemplo:
: clusters esféricos de igual volumen.
: clusters esféricos de distinto volumen.
: clusters elipsoidales de distinto volumen pero misma forma , orientaciones alineadas con el eje.
…
- modelo sin restricciones.
Ahora veremos como ajustar estos modelos. En R hay varios paquetes para ajustar mezclas gaussianas, dos de ellos son mclust y mixtools. También está flexmix
Veamos un ejemplo usando mclust, en este paquete los modelos disponibles son
?mclustModelNames
Mezclas Univariadas
"E" = equal variance (one-dimensional)
"V" = variable variance (one-dimensional)
Mezclas multivariadas
"EII" = spherical, equal volume
"VII" = spherical, unequal volume
"EEI" = diagonal, equal volume and shape
"VEI" = diagonal, varying volume, equal shape
"EVI" = diagonal, equal volume, varying shape
"VVI" = diagonal, varying volume and shape
"EEE" = ellipsoidal, equal volume, shape, and orientation
"EEV" = ellipsoidal, equal volume and equal shape
"VEV" = ellipsoidal, equal shape
"VVV" = ellipsoidal, varying volume, shape, and orientationLa nomenclatura es: E=equal, V=varying, I=identity, y las letras están ordenadas 1ra=volumen, 2a=forma, 3ra=orientación.
En todos los modelos hay parámetros para las probabilidades iniciales mas parámetros para las medias mas los parámetros de la matriz de covarianzas:

Los modelos con menos restricciones tienen más parámetros por estimar y por tanto necesitan de más datos para alcanzar la misma precisión.
Usaremos los datos wine.
library(mclust)
wine <- read.csv("data/wine.csv", stringsAsFactors=FALSE)[, -1]
w_mclust <- Mclust(wine)
summary(w_mclust)
#> ----------------------------------------------------
#> Gaussian finite mixture model fitted by EM algorithm
#> ----------------------------------------------------
#>
#> Mclust VVE (ellipsoidal, equal orientation) model with 3 components:
#>
#> log-likelihood n df BIC ICL
#> -3015.333 178 158 -6849.387 -6850.694
#>
#> Clustering table:
#> 1 2 3
#> 59 69 50Podemos ver más detalles:
summary(w_mclust, parameters = TRUE)
#> ----------------------------------------------------
#> Gaussian finite mixture model fitted by EM algorithm
#> ----------------------------------------------------
#>
#> Mclust VVE (ellipsoidal, equal orientation) model with 3 components:
#>
#> log-likelihood n df BIC ICL
#> -3015.333 178 158 -6849.387 -6850.694
#>
#> Clustering table:
#> 1 2 3
#> 59 69 50
#>
#> Mixing probabilities:
#> 1 2 3
#> 0.3300315 0.3903124 0.2796561
#>
#> Means:
#> [,1] [,2] [,3]
#> Alcohol.content 13.7474607 12.2884420 13.1132188
#> Malic.acid 2.0093958 1.9349628 3.2824045
#> As 2.4524717 2.2414826 2.4395874
#> Alcalinity.in.ash 17.0056425 20.2329719 21.4025951
#> Magnesium 106.2591851 93.9990767 100.0646503
#> Total.phenols 2.8408263 2.2829349 1.6680931
#> Falavanoids 2.9836246 2.1105880 0.7895086
#> Nonflavanoid.phenols 0.2892543 0.3671547 0.4401329
#> Proantocyanins 1.8992977 1.6341503 1.1665817
#> Color.Intensity 5.5361641 3.0977312 7.2299462
#> Hue 1.0619246 1.0596060 0.6915761
#> OD280.OD315 3.1576577 2.8052205 1.6972509
#> Proline 1116.9517345 515.2032715 633.5416560
#>
#> Variances:
#> [,,1]
#> Alcohol.content Malic.acid As
#> Alcohol.content 0.2058329968 -0.004024864 -0.009036939
#> Malic.acid -0.0040248637 0.490091195 0.018910721
#> As -0.0090369389 0.018910721 0.055228146
#> Alcalinity.in.ash -0.0828371420 0.331081359 0.341310246
#> Magnesium 0.1068697208 -0.991646554 0.571126451
#> Total.phenols 0.0052597707 -0.023068454 0.009333359
#> Falavanoids 0.0065310325 -0.026137373 0.016676313
#> Nonflavanoid.phenols 0.0009463792 0.011285445 0.007911553
#> Proantocyanins 0.0149987005 -0.038452295 -0.006737493
#> Color.Intensity 0.1653294086 -0.148232480 -0.026834517
#> Hue 0.0037878962 -0.022582987 0.007238772
#> OD280.OD315 -0.0121794833 0.014950157 0.014614274
#> Proline 17.4628451708 -56.678967719 -3.337718902
#> Alcalinity.in.ash Magnesium Total.phenols
#> Alcohol.content -0.082837142 0.10686972 0.0052597707
#> Malic.acid 0.331081359 -0.99164655 -0.0230684544
#> As 0.341310246 0.57112645 0.0093333589
#> Alcalinity.in.ash 5.971450304 2.01734719 0.0187383181
#> Magnesium 2.017347189 139.89193989 0.9193181647
#> Total.phenols 0.018738318 0.91931816 0.0971100170
#> Falavanoids 0.131007162 0.71138669 0.0842443327
#> Nonflavanoid.phenols 0.044245330 -0.11721737 -0.0061705519
#> Proantocyanins -0.002275193 1.30197911 0.0298378638
#> Color.Intensity -0.255590507 1.28678476 0.0493652310
#> Hue 0.004632895 0.21238589 0.0007562286
#> OD280.OD315 0.168893091 -0.06193507 0.0110876746
#> Proline -52.752038513 732.49780801 13.8868978185
#> Falavanoids Nonflavanoid.phenols Proantocyanins
#> Alcohol.content 0.0065310325 0.0009463792 0.014998700
#> Malic.acid -0.0261373726 0.0112854453 -0.038452295
#> As 0.0166763126 0.0079115530 -0.006737493
#> Alcalinity.in.ash 0.1310071620 0.0442453302 -0.002275193
#> Magnesium 0.7113866941 -0.1172173734 1.301979114
#> Total.phenols 0.0842443327 -0.0061705519 0.029837864
#> Falavanoids 0.1349705248 -0.0069115449 0.062565680
#> Nonflavanoid.phenols -0.0069115449 0.0072891701 -0.007172903
#> Proantocyanins 0.0625656802 -0.0071729033 0.164934604
#> Color.Intensity 0.1144048483 -0.0004530542 0.122825687
#> Hue -0.0006350309 0.0008467983 0.002076420
#> OD280.OD315 0.0226408419 -0.0092570244 0.011252864
#> Proline 10.6322090581 -3.0396915543 23.979311959
#> Color.Intensity Hue OD280.OD315
#> Alcohol.content 0.1653294086 0.0037878962 -0.012179483
#> Malic.acid -0.1482324804 -0.0225829875 0.014950157
#> As -0.0268345172 0.0072387724 0.014614274
#> Alcalinity.in.ash -0.2555905072 0.0046328950 0.168893091
#> Magnesium 1.2867847621 0.2123858908 -0.061935066
#> Total.phenols 0.0493652310 0.0007562286 0.011087675
#> Falavanoids 0.1144048483 -0.0006350309 0.022640842
#> Nonflavanoid.phenols -0.0004530542 0.0008467983 -0.009257024
#> Proantocyanins 0.1228256872 0.0020764204 0.011252864
#> Color.Intensity 1.2827176196 -0.0181024064 -0.063963894
#> Hue -0.0181024064 0.0133148261 -0.001197288
#> OD280.OD315 -0.0639638945 -0.0011972880 0.137646988
#> Proline 89.6000335505 8.4888492803 -8.638897429
#> Proline
#> Alcohol.content 17.462845
#> Malic.acid -56.678968
#> As -3.337719
#> Alcalinity.in.ash -52.752039
#> Magnesium 732.497808
#> Total.phenols 13.886898
#> Falavanoids 10.632209
#> Nonflavanoid.phenols -3.039692
#> Proantocyanins 23.979312
#> Color.Intensity 89.600034
#> Hue 8.488849
#> OD280.OD315 -8.638897
#> Proline 48054.698849
#> [,,2]
#> Alcohol.content Malic.acid As
#> Alcohol.content 0.290957199 0.041494976 -0.015172597
#> Malic.acid 0.041494976 0.959616485 0.031392560
#> As -0.015172597 0.031392560 0.100832278
#> Alcalinity.in.ash -0.139222786 0.557508416 0.664954718
#> Magnesium -0.151719216 -0.652094100 1.080065801
#> Total.phenols -0.040256458 -0.002113848 0.029119508
#> Falavanoids -0.060147746 -0.004874673 0.039501254
#> Nonflavanoid.phenols 0.007548535 0.015915807 0.013514996
#> Proantocyanins -0.040749045 -0.009251307 -0.006148156
#> Color.Intensity 0.125146552 -0.080664399 -0.033632185
#> Hue 0.004211772 -0.034179000 0.007228388
#> OD280.OD315 -0.042241674 0.026423741 0.028446620
#> Proline 8.626596290 -27.985876625 -1.659845775
#> Alcalinity.in.ash Magnesium Total.phenols
#> Alcohol.content -0.13922279 -0.15171922 -0.040256458
#> Malic.acid 0.55750842 -0.65209410 -0.002113848
#> As 0.66495472 1.08006580 0.029119508
#> Alcalinity.in.ash 11.66688422 4.59938073 0.047875643
#> Magnesium 4.59938073 234.48630651 1.361037876
#> Total.phenols 0.04787564 1.36103788 0.264992918
#> Falavanoids 0.25897013 1.05405977 0.257799261
#> Nonflavanoid.phenols 0.08314979 -0.14875746 -0.022078478
#> Proantocyanins 0.01893329 1.84347909 0.127681628
#> Color.Intensity -0.40238687 0.53161273 0.007950886
#> Hue 0.02452953 0.21105543 -0.002551795
#> OD280.OD315 0.31725455 0.05715343 0.110329268
#> Proline -26.09488891 359.20264305 6.843821755
#> Falavanoids Nonflavanoid.phenols Proantocyanins
#> Alcohol.content -0.060147746 0.007548535 -0.040749045
#> Malic.acid -0.004874673 0.015915807 -0.009251307
#> As 0.039501254 0.013514996 -0.006148156
#> Alcalinity.in.ash 0.258970125 0.083149785 0.018933288
#> Magnesium 1.054059766 -0.148757464 1.843479090
#> Total.phenols 0.257799261 -0.022078478 0.127681628
#> Falavanoids 0.402859513 -0.030553714 0.219205900
#> Nonflavanoid.phenols -0.030553714 0.013721881 -0.025794233
#> Proantocyanins 0.219205900 -0.025794233 0.397162067
#> Color.Intensity 0.047959574 0.003634641 0.055879900
#> Hue -0.004842160 0.001549258 -0.003299242
#> OD280.OD315 0.160712163 -0.021481045 0.121953613
#> Proline 5.239795551 -1.499604179 11.823144132
#> Color.Intensity Hue OD280.OD315
#> Alcohol.content 0.125146552 0.004211772 -0.042241674
#> Malic.acid -0.080664399 -0.034179000 0.026423741
#> As -0.033632185 0.007228388 0.028446620
#> Alcalinity.in.ash -0.402386869 0.024529529 0.317254548
#> Magnesium 0.531612726 0.211055426 0.057153426
#> Total.phenols 0.007950886 -0.002551795 0.110329268
#> Falavanoids 0.047959574 -0.004842160 0.160712163
#> Nonflavanoid.phenols 0.003634641 0.001549258 -0.021481045
#> Proantocyanins 0.055879900 -0.003299242 0.121953613
#> Color.Intensity 1.025931508 -0.020505202 -0.072269929
#> Hue -0.020505202 0.041007971 -0.002452333
#> OD280.OD315 -0.072269929 -0.002452333 0.215689168
#> Proline 44.247495205 4.190420761 -4.267192790
#> Proline
#> Alcohol.content 8.626596
#> Malic.acid -27.985877
#> As -1.659846
#> Alcalinity.in.ash -26.094889
#> Magnesium 359.202643
#> Total.phenols 6.843822
#> Falavanoids 5.239796
#> Nonflavanoid.phenols -1.499604
#> Proantocyanins 11.823144
#> Color.Intensity 44.247495
#> Hue 4.190421
#> OD280.OD315 -4.267193
#> Proline 23730.772720
#> [,,3]
#> Alcohol.content Malic.acid As
#> Alcohol.content 0.364795175 0.064640215 -0.014255292
#> Malic.acid 0.064640215 1.202742989 0.009619805
#> As -0.014255292 0.009619805 0.034969282
#> Alcalinity.in.ash -0.035993225 0.182864164 0.269439543
#> Magnesium -0.090609764 -0.365698545 0.624920131
#> Total.phenols 0.031616437 -0.055151917 0.023026263
#> Falavanoids 0.083433862 -0.103556511 -0.009829708
#> Nonflavanoid.phenols 0.004430543 0.021256335 0.001934607
#> Proantocyanins 0.077373081 -0.072496459 -0.000510812
#> Color.Intensity 0.769007331 -0.245044458 -0.061093951
#> Hue -0.017715605 -0.035787308 0.004035577
#> OD280.OD315 -0.023346744 -0.013926442 0.013095158
#> Proline 4.869717266 -15.801209810 -0.937506525
#> Alcalinity.in.ash Magnesium Total.phenols
#> Alcohol.content -0.035993225 -0.09060976 0.031616437
#> Malic.acid 0.182864164 -0.36569855 -0.055151917
#> As 0.269439543 0.62492013 0.023026263
#> Alcalinity.in.ash 4.687651663 2.69982780 0.034940535
#> Magnesium 2.699827797 134.94975622 0.783495550
#> Total.phenols 0.034940535 0.78349555 0.112711973
#> Falavanoids 0.135032979 0.60843506 0.005760379
#> Nonflavanoid.phenols 0.032945039 -0.08524479 0.003875283
#> Proantocyanins 0.034275071 1.06047025 0.047307104
#> Color.Intensity 0.022939765 0.29043216 0.184482532
#> Hue 0.005182554 0.12098019 0.000579047
#> OD280.OD315 0.123688178 0.03632290 -0.003086967
#> Proline -14.738807160 202.78506111 3.863769830
#> Falavanoids Nonflavanoid.phenols Proantocyanins
#> Alcohol.content 0.083433862 4.430543e-03 7.737308e-02
#> Malic.acid -0.103556511 2.125633e-02 -7.249646e-02
#> As -0.009829708 1.934607e-03 -5.108120e-04
#> Alcalinity.in.ash 0.135032979 3.294504e-02 3.427507e-02
#> Magnesium 0.608435055 -8.524479e-02 1.060470e+00
#> Total.phenols 0.005760379 3.875283e-03 4.730710e-02
#> Falavanoids 0.179195550 -5.819478e-03 6.602239e-02
#> Nonflavanoid.phenols -0.005819478 1.605783e-02 -4.840697e-05
#> Proantocyanins 0.066022386 -4.840697e-05 1.223608e-01
#> Color.Intensity 0.584663452 2.722327e-02 4.906441e-01
#> Hue -0.016193650 -1.250589e-03 -1.000528e-02
#> OD280.OD315 -0.016412517 -6.006693e-03 -1.510007e-02
#> Proline 2.957297389 -8.467936e-01 6.674755e+00
#> Color.Intensity Hue OD280.OD315
#> Alcohol.content 0.76900733 -0.017715605 -0.023346744
#> Malic.acid -0.24504446 -0.035787308 -0.013926442
#> As -0.06109395 0.004035577 0.013095158
#> Alcalinity.in.ash 0.02293976 0.005182554 0.123688178
#> Magnesium 0.29043216 0.120980192 0.036322896
#> Total.phenols 0.18448253 0.000579047 -0.003086967
#> Falavanoids 0.58466345 -0.016193650 -0.016412517
#> Nonflavanoid.phenols 0.02722327 -0.001250589 -0.006006693
#> Proantocyanins 0.49064410 -0.010005284 -0.015100071
#> Color.Intensity 5.78108574 -0.166574834 -0.231453078
#> Hue -0.16657483 0.015609471 0.007313253
#> OD280.OD315 -0.23145308 0.007313253 0.103382839
#> Proline 24.97418230 2.366371149 -2.409193145
#> Proline
#> Alcohol.content 4.8697173
#> Malic.acid -15.8012098
#> As -0.9375065
#> Alcalinity.in.ash -14.7388072
#> Magnesium 202.7850611
#> Total.phenols 3.8637698
#> Falavanoids 2.9572974
#> Nonflavanoid.phenols -0.8467936
#> Proantocyanins 6.6747551
#> Color.Intensity 24.9741823
#> Hue 2.3663711
#> OD280.OD315 -2.4091931
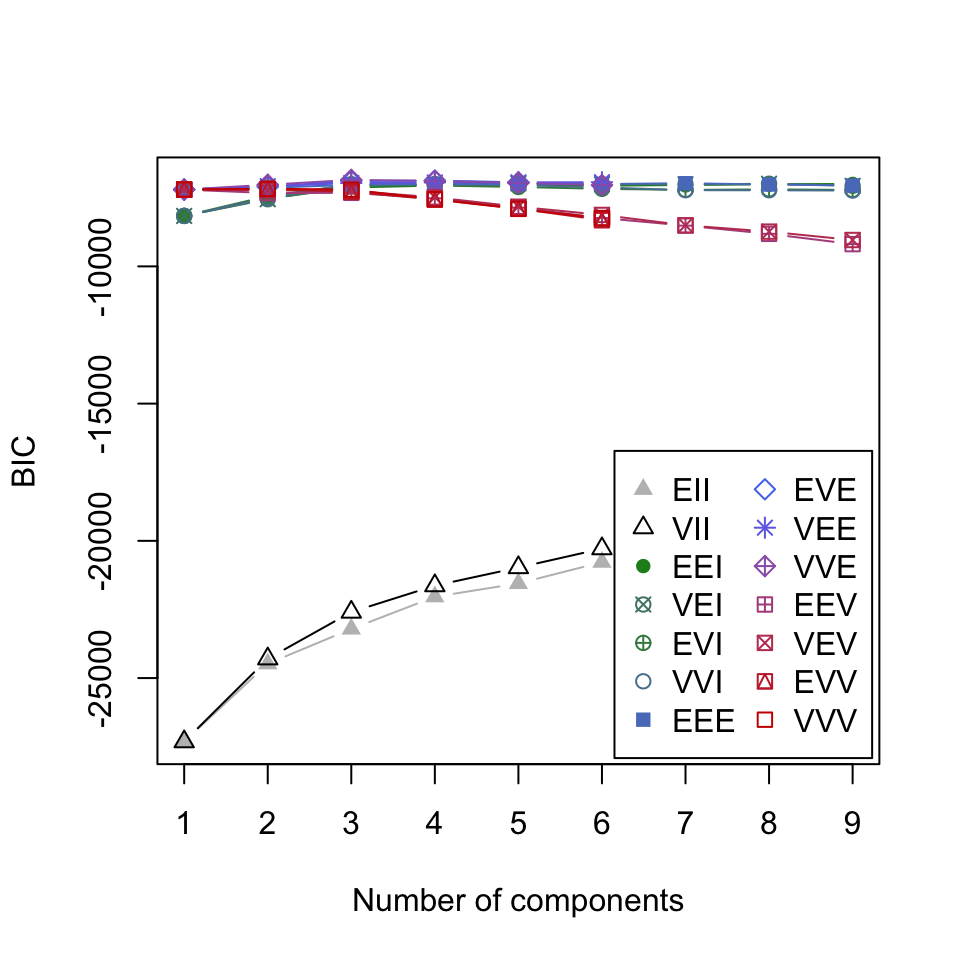
#> Proline 13399.5878181Y podemos ver una tabla con el BIC para cada modelo, es importante tomar en cuenta que en mclust el BIC se define como:
es el número de parámetros y el número de observaciones. Por tanto, buscamos maximizar el BIC.
round(w_mclust$BIC)
#> Bayesian Information Criterion (BIC):
#> EII VII EEI VEI EVI VVI EEE EVE VEE VVE EEV
#> 1 -27318 -27318 -8161 -8161 -8161 -8161 -7201 -7201 -7201 -7201 -7201
#> 2 -24478 -24297 -7534 -7553 -7440 -7496 -7138 -7099 -7077 -7027 -7328
#> 3 -23210 -22595 -7125 -7065 -7024 -7003 -7026 -6889 -6963 -6849 -7321
#> 4 -22037 -21633 -7055 -7014 -7053 -6993 -7013 -6874 -6943 -6886 -7520
#> 5 -21552 -20974 -7020 -6975 -7107 -7041 -6992 -6937 -6938 -6957 -7860
#> 6 -20777 -20285 -7061 -7006 -7175 -7122 -7000 NA -6933 -7039 -8236
#> 7 -20584 -19978 -7036 -7014 -7214 -7223 -6969 NA NA NA -8514
#> 8 -20511 -19167 -7009 -6988 -7194 -7227 -7018 NA NA NA -8815
#> 9 -19054 -18784 -6995 -7065 -7215 -7236 -7052 NA NA NA -9188
#> VEV EVV VVV
#> 1 -7201 -7201 -7201
#> 2 -7229 -7181 -7169
#> 3 -7250 -7285 -7204
#> 4 -7484 -7563 -7555
#> 5 -7826 -7895 -7905
#> 6 -8125 -8314 -8245
#> 7 -8503 NA NA
#> 8 -8734 NA NA
#> 9 -9039 NA NA
#>
#> Top 3 models based on the BIC criterion:
#> VVE,3 EVE,4 VVE,4
#> -6849 -6874 -6886
w_mclust
#> 'Mclust' model object: (VVE,3)
#>
#> Available components:
#> [1] "call" "data" "modelName" "n"
#> [5] "d" "G" "BIC" "bic"
#> [9] "loglik" "df" "hypvol" "parameters"
#> [13] "z" "classification" "uncertainty"En la tabla de arriba, los renglones indican el número de conglomerados.
El paquete incluye algunas gráficas auxiliares.
Y podemos hacer otras gráficas, por ejemplo podemos usar tourr para explorar los datos multivariados.
library(tourr)
w_mclust <- Mclust(wine, G = 3, modelNames = "VVI")
summary(w_mclust)
cl_p <- data.frame(cluster = w_mclust$classification, wine)
aps <- 2
fps <- 20
mat <- rescale(wine)
tour <- new_tour(mat, grand_tour(), NULL)
start <- tour(0)
proj_data <- reactive({
invalidateLater(1000 / fps, NULL);
step <- tour(aps / fps)
data.frame(center(mat %*% step$proj),
clusters = factor(w_mclust$classification))
})
proj_data %>% ggvis(~X1, ~X2, fill = ~clusters) %>%
layer_points() %>%
scale_numeric("x", domain = c(-1, 1)) %>%
scale_numeric("y", domain = c(-1, 1)) %>%
set_options(duration = 0)6.3.1 Observaciones
Las mezclas Gaussianas son sensibles a datos atípicos (outliers).
Puede haber inestabilidad si los componentes Gaussianos no están separados apropiadamente o si los datos son chicos.
A comparación de otros métodos de clustering el tener un modelo probabilístico explícito hace posible verificar supuestos y tener medidas de ajuste. Por ejemplo, podemos usar BIC para elegir el número de clusters.
k-medias es un caso particular de mezclas gaussianas donde en cada paso se realizan asignaciones duras.
Desventajas: los resultados dependen de la inicialización del algoritmo EM, puede ser demasiado flexible o inestable.
Las mezclas gaussianas se pueden extender a mezclas de modelos de regresión que pueden ser modelos lineales estándar o modelos lineales generalizados. Esto esta implementado en el paquete flexmix que utiliza el algoritmo EM.
6.3.2 Aplicación: Background Subtraction
Background Subtraction es una técnica de procesamiento de imágenes donde se buscan extraer los objetos en el frente de la imagen. Por ejemplo, puede ser de interés detectar autos, animales o personas de una imagen. Ejemplos:
Monitoreo de tráfico (contar vehiculos, detectar y seguir vehículos).
Reconocimiento de actividades humanas (correr, caminar, brincar).
Seguimiento de objetos (seguir la bola en el tenis).

Los modelos de mezclas gaussianas son populares para esta tarea: la idea básica es suponer que la intensidad de cada pixel en una imagen se puede modelar usando un modelo de mezclas Gaussianas. Después se determina que intensidades corresponden con mayor probabilidad al fondo seleccionando como fondo las gaussianas con menos varianza y mayor evidencia ().
En OpencCV hay tres algoritmos implementados para sustraer fondo, estos usan variantes de mezclas gaussianas: