4.2 Matriz de precisión
Los elementos de la diagonal de \(D=\Sigma^{-1}\) pueden interpretarse de la siguiente manera:
- Los elementos de la diagonal están relacionados con la \(R^2\) de la regresión de la variable correspondiente con respecto a todas las demás: \[D_{ii}= 1/(1-R_i^2)\]
fit_alg <- lm(ALG ~ MECH + VECT + ANL + STAT, data = marks_s)
# summary(fit_alg)
R2 <- summary(fit_alg)$r.squared
D_alg <- 1 / (1 - R2)
fit_anl <- lm(ANL ~ MECH + VECT + ALG + STAT, data = marks_s)
R2 <- summary(fit_anl)$r.squared
D_anl <- 1 / (1 - R2)
c(D_alg, D_anl)
#> [1] 3.042821 2.178002- Los elementos fuera de la diagonal están relacionados con la correlación parcial entre dos variables. La correlación parcial \(\rho_{ij|resto}\) entre \(X_i\) y \(X_j\) es la correlación entre los residuales de la regresión de \(X_i\) contra el resto de las variables y los residuales de \(X_j\) contra el resto de las variables. Se puede interpretar como la correlación que existe entre \(X_i\) y \(X_j\) cuando mantenemos el resto de las variables fijas. Cuando este coeficiente es mucho más chico que la correlación usual entre \(X_i\) y \(X_j\) quiere decir que una buena parte de la correlación de estas variables puede explicarse debido a variación entre el resto de las variables. Específicamente, \[\rho_{ij|resto}-\frac{D_{ij}}{\sqrt{D_{ii}D_{jj}}}.\]
fit_alg_2 <- lm(ALG ~ MECH + VECT + STAT, data = marks_s)
fit_anl_2 <- lm(ANL ~ MECH + VECT + STAT, data = marks_s)
cor(residuals(fit_alg_2), residuals(fit_anl_2))
#> [1] 0.4318565
- D[3, 4] / sqrt(D[3, 3] * D[4, 4])
#> [1] 0.4318565Notemos que no es lo mismo que la correlación usual entre STAT y MECH.
Podemos examinar todas las correlaciones parciales haciendo:
D <- solve(cor(marks_s))
parciales_D <- -(t(D * (1/sqrt(diag(D)))) * (1 / sqrt(diag(D))))
round(parciales_D, 2)
#> MECH VECT ALG ANL STAT
#> MECH -1.00 0.33 0.23 0.00 0.02
#> VECT 0.33 -1.00 0.28 0.08 0.02
#> ALG 0.23 0.28 -1.00 0.43 0.36
#> ANL 0.00 0.08 0.43 -1.00 0.25
#> STAT 0.02 0.02 0.36 0.25 -1.00 En cada una de las imágenes de abajo
indica si la estructura de la gráfica del lado derecho corresponde a la matriz
de correlación parcial (cuadro indica valor distinto a cero y vacío indica
cero).
En cada una de las imágenes de abajo
indica si la estructura de la gráfica del lado derecho corresponde a la matriz
de correlación parcial (cuadro indica valor distinto a cero y vacío indica
cero).
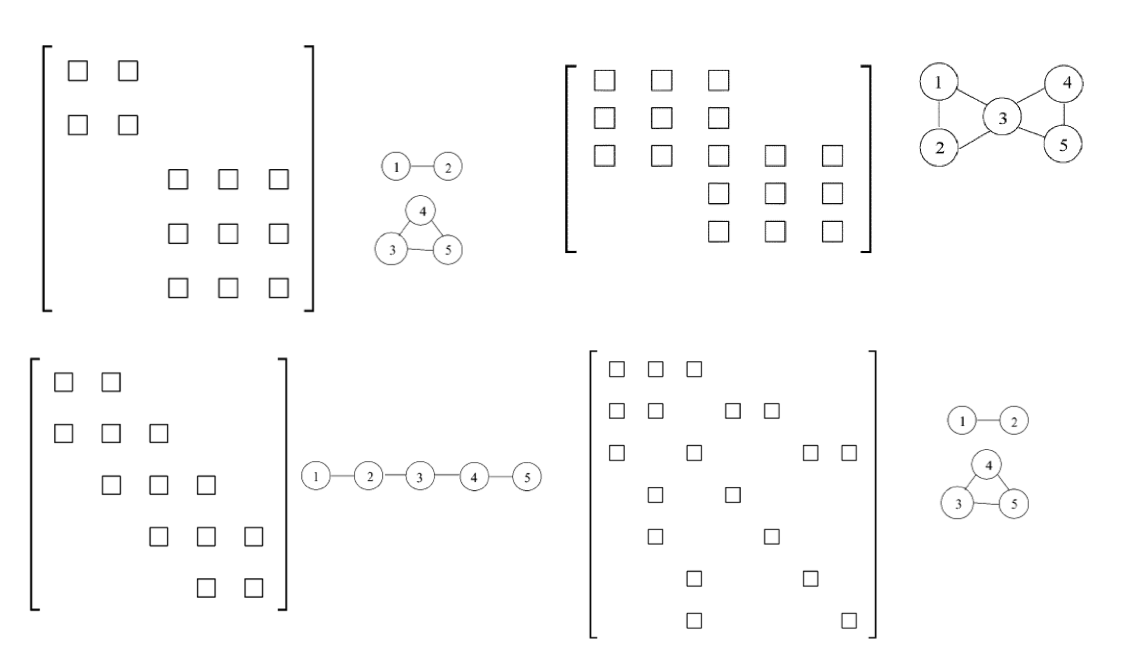 a. Arriba izquierda: Verdadero
a. Arriba izquierda: Verdadero
b. Arriba derecha: Verdadero
c. Abajo izquierda: Verdadero
d. Abajo derecha: Verdadero
4.2.1 Independencia condicional para la normal multivariada
De la discusión anterior, vemos que cuando \((X_1,\ldots, X_k)\) es normal multivariada, entonces \(X_i\) es independiente de \(X_j\) dado el resto de las variables si y sólo si la correlación parcial es cero, o de otra forma, cuando \(D_{ij}=0\).
Podemos ahora apelar a nuestros resultados anteriores (propiedades de Markov por pares, global y distribución de Gibbs) para demostrar el siguiente teorema (aunque también se puede resolver mediante cálculo):
Si \(X=(X_a,X_b, X_c)\) es normal multivariada, donde \(X_a,X_b,X_c\) son bloques de variables. Los vectores \(X_a\) y \(X_b\) son condicionalmente independientes dado \(X_c\) si y sólo si el bloque \(D_{ab}\) de la precisión o varianza inversa \(D=\Sigma^{-1}\) es igual a cero.
round(cov(marks))
#> MECH VECT ALG ANL STAT
#> MECH 306 127 102 106 117
#> VECT 127 173 85 95 99
#> ALG 102 85 113 112 122
#> ANL 106 95 112 220 156
#> STAT 117 99 122 156 298Si estandarizamos las variables para que la variabilidad sea comparable, obtenemos la matriz
cor(marks)
#> MECH VECT ALG ANL STAT
#> MECH 1.0000000 0.5534052 0.5467511 0.4093920 0.3890993
#> VECT 0.5534052 1.0000000 0.6096447 0.4850813 0.4364487
#> ALG 0.5467511 0.6096447 1.0000000 0.7108059 0.6647357
#> ANL 0.4093920 0.4850813 0.7108059 1.0000000 0.6071743
#> STAT 0.3890993 0.4364487 0.6647357 0.6071743 1.0000000Cuya inversa es
inv_1 <- round(solve(cor(marks)), 2)
inv_1
#> MECH VECT ALG ANL STAT
#> MECH 1.60 -0.56 -0.51 0.00 -0.04
#> VECT -0.56 1.80 -0.66 -0.15 -0.04
#> ALG -0.51 -0.66 3.04 -1.11 -0.86
#> ANL 0.00 -0.15 -1.11 2.18 -0.52
#> STAT -0.04 -0.04 -0.86 -0.52 1.92Donde vemos los coeficientes de esta inversa para los pares ANL-MECH, ANL-VECT, VEC-STAT, MECH-STAT son cercanos a cero. Esto implica que son variables cercanas a la independencia dado el resto de las variables. Así que un modelo apropiado para estos datos tiene todas las aristas, excepto la lista que mencionó arriba. Terminamos entonces con la gráfica
library(Rgraphviz)
#> Loading required package: graph
#> Loading required package: BiocGenerics
#> Loading required package: parallel
#>
#> Attaching package: 'BiocGenerics'
#> The following objects are masked from 'package:parallel':
#>
#> clusterApply, clusterApplyLB, clusterCall, clusterEvalQ,
#> clusterExport, clusterMap, parApply, parCapply, parLapply,
#> parLapplyLB, parRapply, parSapply, parSapplyLB
#> The following objects are masked from 'package:bnlearn':
#>
#> path, score
#> The following objects are masked from 'package:dplyr':
#>
#> combine, intersect, setdiff, union
#> The following objects are masked from 'package:stats':
#>
#> IQR, mad, sd, var, xtabs
#> The following objects are masked from 'package:base':
#>
#> anyDuplicated, append, as.data.frame, basename, cbind,
#> colnames, dirname, do.call, duplicated, eval, evalq, Filter,
#> Find, get, grep, grepl, intersect, is.unsorted, lapply, Map,
#> mapply, match, mget, order, paste, pmax, pmax.int, pmin,
#> pmin.int, Position, rank, rbind, Reduce, rownames, sapply,
#> setdiff, sort, table, tapply, union, unique, unsplit, which,
#> which.max, which.min
#>
#> Attaching package: 'graph'
#> The following objects are masked from 'package:bnlearn':
#>
#> degree, nodes, nodes<-
#> The following object is masked from 'package:stringr':
#>
#> boundary
#> Loading required package: grid
data(marks)
ug_1 <- ug(~ ANL:STAT + ALG:STAT + ALG:ANL + MECH:ALG + VECT:ALG + VECT:MECH)
plot(ug_1)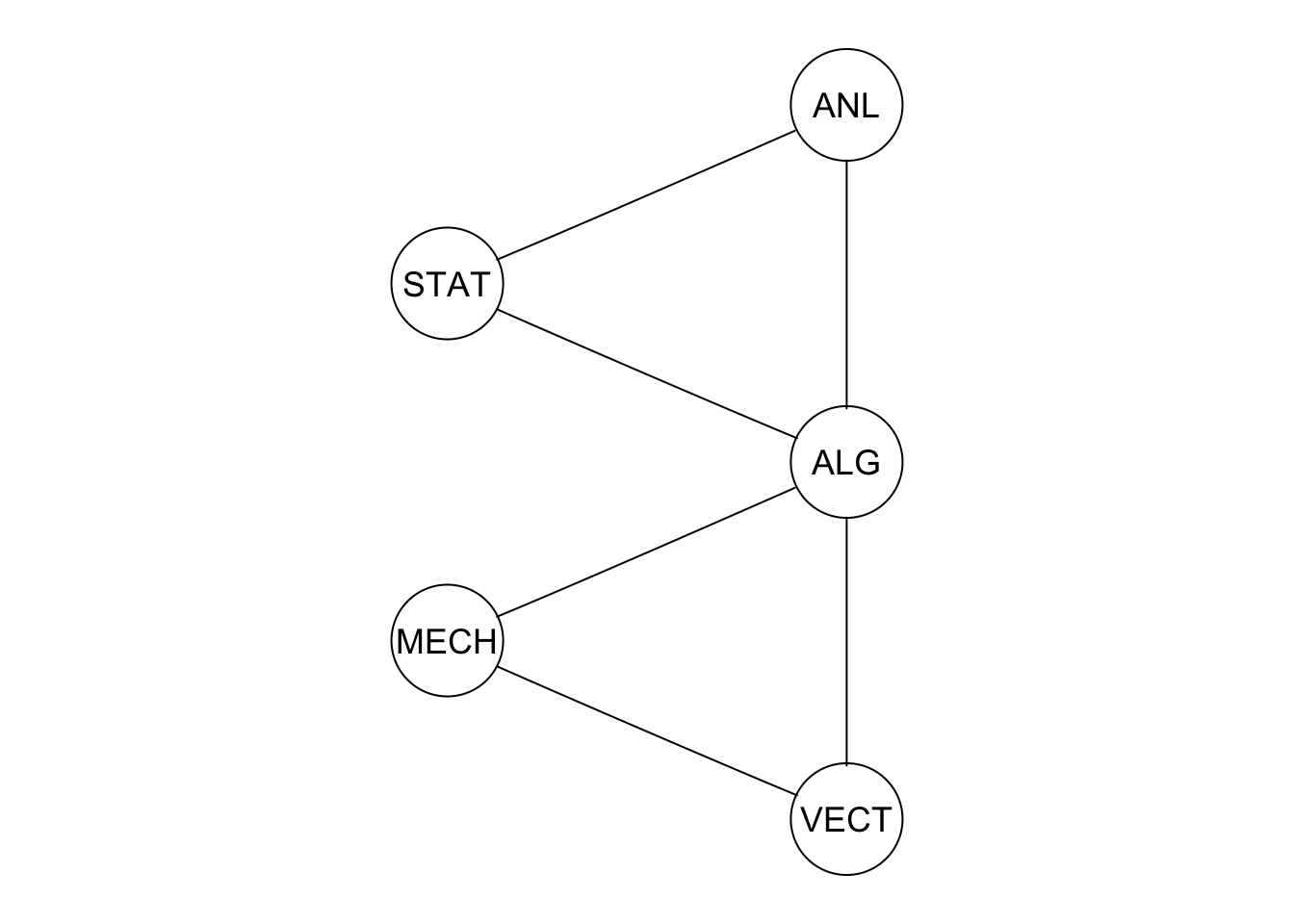
Podemos graficar mostrando las correlaciones parciales:
nombres <- sapply(edgeList(ug_1), function(x){
nombre <- paste0('', paste(c(x[1], x[2]), collapse = "~"), '')
nombre
})
peso <- sapply(edgeList(ug_1), function(x){
peso <- as.character(round(parciales_D[x[1], x[2]], 2))
})
etiquetas <- peso
names(etiquetas) <- nombres
Rgraphviz::plot(ug_1, edgeAttrs = list(label = etiquetas))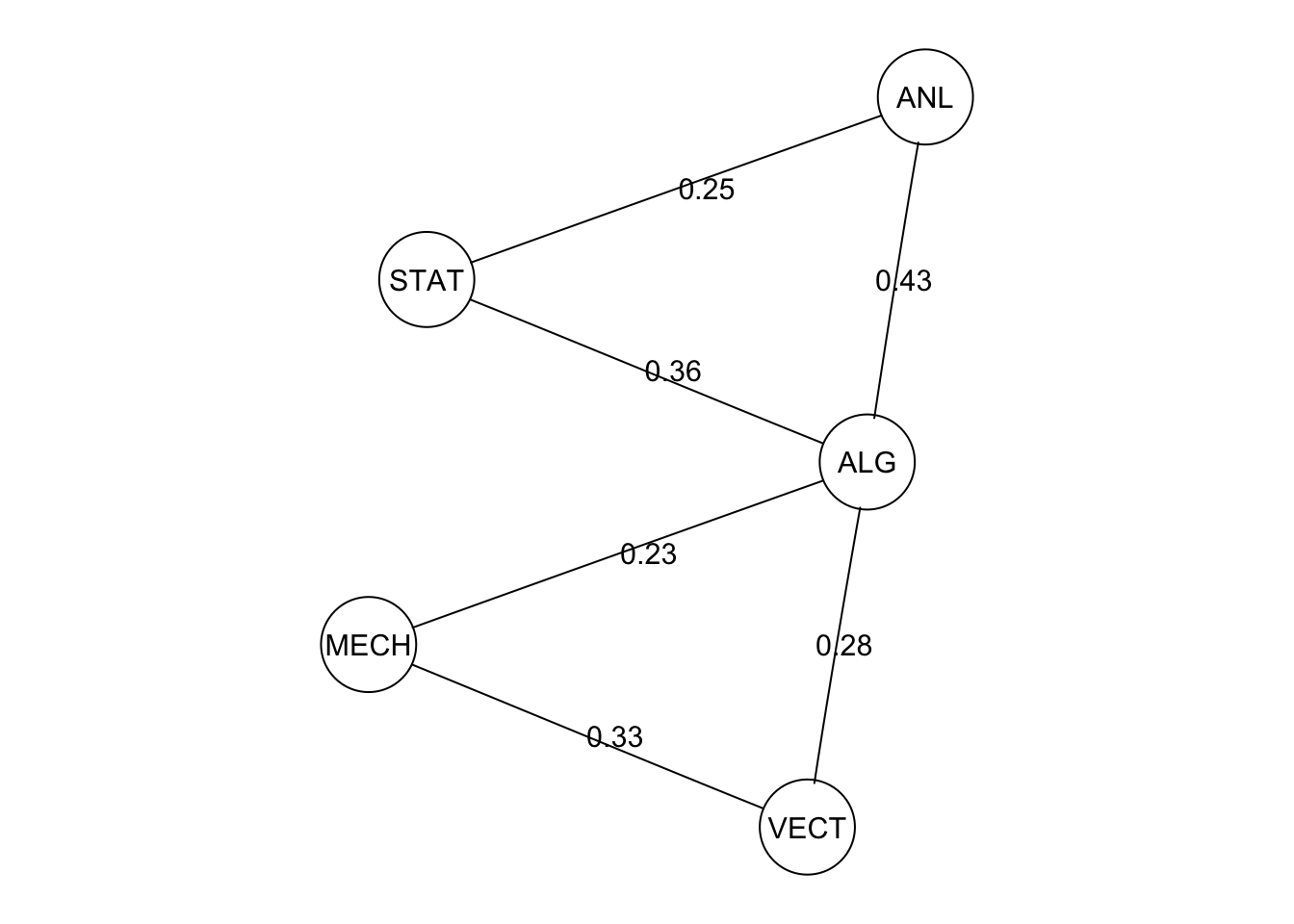
4.2.2 Estimación de máxima verosimilitud con estructura conocida
Un modelo no dirigido gaussiano especifica una distribución normal bivariada
\(N(\mu, \Sigma)\) tal que
* Si no hay una arista entre \(X_i\) y \(X_j\), entonces \(D_{ij}=D_{ji}=0\)
donde \(D=\Sigma^{-1}\).
De esta forma, vemos que cuando tenemos una estructura no dirigida dada, nuestro trabajo es estimar la matriz de covarianza (o la precisión) con restricciones de ceros dadas por las independencias condicionales.
Podemos entonces maximizar la verosimilitud con las restricciones sobre la inversa de la matriz de varianza y covarianza. En general no es problema trivial.